トップページ > 研究組織一覧 > 分野・独立ユニットグループ > 希少がん研究分野 > 希少がんを研究するということ
希少がんを研究するということ
1.はじめに:希少がんは希少ではない
希少がんは、「新規に診断される症例の数が10万人あたり年間6例未満のがん」と定義されている。すなわち、希少がんとは、発症数によって定義されるがんである。メジャーがんである肺がんが、新規に診断される症例数は10万人あたり年間200人程度であることを考えると、希少がんの希少性がご理解いただけるだろうか。がんは一般に、由来する臓器、組織型、分子背景などを基に分類される。「発症数」という個々の腫瘍の生物学的特性とはまったく異なる切り口で定義される希少がんは、その構成がユニークである。すなわち、希少がんには200種類近くもの異なるがんが含まれており、その内訳は、肉腫、悪性脳腫瘍、皮膚腫瘍、眼腫瘍、中皮腫、副腎がん、神経内分泌腫瘍など実に多岐にわたる。
個々の希少がんの症例数は「新規に診断される症例の数が10万人あたり年間6例未満」と少ないのだが、そのような希少ながんがたくさん集まった結果として、希少がん全体の症例は意外にも多い。ヨーロッパで新規に希少がんと診断される患者はがん患者全体の約22%にもおよぶ[1]。本邦では約15%、数にして約10万人の方が新規に希少がんと診断されている[2]。すなわち、希少がんを患う患者の数は、たいていのがんの患者よりも多いのである(図1)。
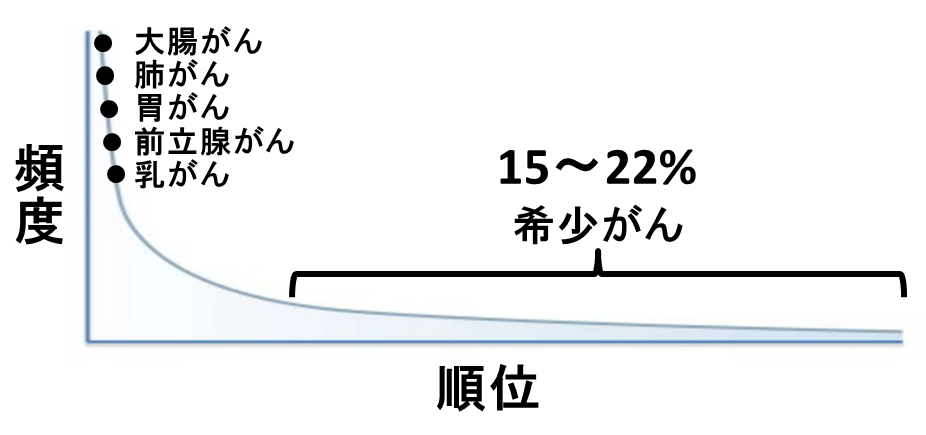
図1:がんのロングテール。頻度の高い少数のがんと、頻度の低い多数のがんが存在する。全体的には「希少がん」はその定義と裏腹に、実は希少ではない。
「希少がんの研究」というと重箱の隅をつつくようなマイナーな研究だと思われるかもしれないが、実はそうではない。希少がんの研究とは、多くの患者を対象とする重大かつ壮大な研究なのである。希少がんの研究には複眼的な視点が必要である。症例数が少ないということ以外に共通項がないがんを扱うのが希少がんの研究なので、従来のがん研究の枠にとらわれない視点が求められる。たとえば、肉腫と悪性脳腫瘍と眼腫瘍と神経内分泌腫瘍を同じ研究者が研究するということは、従来のがん研究ではなかなかない状況である。しかし、希少がん研究ではそれらに共通する課題をみつけて取り組む必要がある。問題設定から解決までをすべて考えて行うというのは研究者として当然といえば当然の行為なのだが、従来のがんの分類とは異なる視点が必要とされるという意味で、希少がんの研究はユニークである。
「希少ながんは希少ではない」とはいえ、それは希少がん全体を見渡した場合であって、個々の希少ながんの症例数は少ない。希少ながんを個別的にみていくと、そこには症例が少ないことに起因する独特の問題が存在する。まず、希少ながんの研究はがん研究の主流ではなく、どちらかといえばマイナーである。希少ながんを研究する研究者の数は少なく、使われてきた研究費も多くない(「第3次対がん総合戦略研究事業の全体報告と、がん研究の今後のあり方について」(PDF:7,180KB))。新しい解析技術はメジャーがんの研究にまず導入され、いろいろなアイデアや可能性が試され、欠点が洗い出され、技術として徹底的に使いこまれてから、ごく一部の希少ながんに応用される。しっかり技術開発が進んでからの応用なので無駄がなく、普及してからの応用なので効率がよいともいえる。しかし、希少ながんの研究が遅れがちになっても不思議ではない。また、最先端の解析技術はあらゆる希少ながんの研究に導入されるわけでなく、肉腫や悪性脳腫瘍など、希少ながんの中でも症例数が比較的多いものに限定されて使われている。200種類近くある希少ながんの最後の一つに最先端の技術が応用されるころには、その技術はもはや最先端ではなくなっているだろう。
臨床的には、希少であることに由来するさまざまな医療上・受療上の課題が存在する。患者さんにとっては、どこにいけば適切な治療を受けることができるのかがわからないというのが深刻な問題である。希少ながんの情報は一般の方にとって入手やすいとは言えない。医療従事者にとっては、生涯に一度も遭遇しなくてもおかしくないほど希ながんの患者が外来にある日突然現れても、適格に診断・治療を施すことができないし、どこに紹介してよいかわからないかもしれない。病理診断についても同様である。治療法の拠り所となる病理診断がそもそも不確かだと、エビデンスに裏付けられた標準的な治療法が存在したとしても適格に適応できない。さらに、症例が少ないことから十分な症例数を元に臨床試験を実施することはきわめて困難なので、新しい治療法の開発はふつう以上に難しい。市場規模が小さいこともあって、希少ながんの臨床研究には独特の難しさがつきまとう。
希少であることに由来するこのような問題を希少がんのすべてが抱えているわけではない。症例数が少なくても、診断・治療法が確立されており特定の専門家が診療にあたっているような希少がんは、希少だからという理由で新しい取り組みが必要というわけではない。一方、手術以外に科学的根拠に基づく治療法が乏しい希少がんは、特に注力して取り組む必要がある。
2.希少がんへの国立がん研究センターの取り組み
希少がんのこのような問題は今に始まったことではない。しかし、肉腫が「忘れられたがん」と言われるように、希少がんの問題はあまり注目されることはなかった。ロングテールの尻尾が見落とされるのはよくあることで、医療も同じなのだろう。しかし、2014年に打ち出された「がん研究10か年戦略」では、具体的研究事項として、「希少がん等に関する研究」も重点研究領域の一つになった。そして、国立がん研究センターも希少がんの問題の重要性を鑑み、2014年に「希少がんセンター」を立ち上げ、異なる科の医療従事者が一体となって希少がんの診療に対応する体制の構築を始めた。希少がんの診療には複数の科の臨床医が携わっており、臨床科の枠を越えた連携は重要である。また、「希少がんセンター」ではホットラインを設定し、患者やその家族からの電話での問い合わせに専任のスタッフが応えている。厚生労働省も2014年に「希少がん医療・支援のあり方に関する検討会」を設置し、希少がんの定義や希少がん医療のあり方について検討を開始した。
このような病院や国の取り組みに対応して、国立がん研究センター研究所でも「希少がん研究分野」を2014年に設立した。希少ながんの研究は個々の研究者が以前から取り組んでいたのだが、一つの研究単位として扱うことになった。希少ながんの一つである肉腫のプロテオーム解析を10数年行なっていた経緯から、私がそのPI(Principal Investigator)を勤めることになった。「希少がん研究」というくくりは基礎研究の分野では私の知る限りではかつて存在せず、「希少がん」を標榜する研究室は国内には存在しなかった。もっとも、私は特定の希少ながんを集中的に研究していたわけではないし、希少ながんの患者さんを診たことはない。プロテオミクスなどの技術を軸に、腫瘍の種類にはこだわらずに広くがんを研究してきた。特に興味をもっている遺伝子・タンパク質もなく、どちらかといえば技術系の研究者である。今までに、肺がん、大腸がん、胃がん、肝細胞癌、食道がん、などいろいろながんのプロテオームを調べてきた。どのがんにも共通した課題や分子背景が存在すると同時に、個々のがんに特有の問題が存在することに気づかされる。そのような経験から、200種類近いがんを扱う希少がん研究はたいへん興味深いと思っている。「希少がんを研究するとはどういうことなのか」、「従来からある肉腫や悪性脳腫瘍など個々の希少ながんの研究と、『希少がんを研究すること』はどう違うのか」など、研究の根本を考えるところから研究を開始した。さまざまな希少ながんに共通に存在する問題を考えて、希少がんの研究基盤の構築、個別の希少ながんの新しい治療法を目指した研究に取り組むことにした。また、希少がん研究で培ったノウハウをがん研究一般に応用する「リバース・イノベーション」も目標としている。以下に取り組みの一端をご紹介する。
3.希少がんを研究するということ
「希少ながんはなぜ希少なのか」という研究テーマが、基礎研究の視点からは本質的に興味深い。希少ながんはそれぞれの事情があって希少になっていると考えられる。「希少ながんはなぜ希少なのか」という疑問は学術的な好奇心に留まらず、研究のアプローチを最適化したり、新しい生物学を切り開いたり、臨床応用を考えたりするためにも重要である。
まず、特定のがんを引き起こすイベントが極めて希な場合、そのがんは希少ながんになる。たとえば、約3分の1の肉腫では融合遺伝子が臨床的・病理学的に独特の病態を導いている。しかし、融合遺伝子があれば肉腫が発生するというものではなく、特定の細胞の発生のあるタイミングで融合遺伝子の異常が発生することがポイントのようである。このように、発がんの原因となる遺伝子の数が少数で、その少数の遺伝子にたまたま特定の変異が特定のタイミングで特定の細胞に発生する確率はきわめて少ない場合には、そのがんは希少ながんになる。この現象を臨床的にうまく利用することができるかもしれない。たとえば、原因となる少数の遺伝子の異常は鑑別診断のよいバイオマーカーになりうるし、その遺伝子の機能を阻害する手段は特異性が高く副作用が少ない治療法になりうる。ある医学研究が成功するとは、特定の疾患においてほとんどの症例に有効な診断法や治療法が確立されることだろう。原因となる遺伝子の異常がきわめて限られているから希少になっているがんは、その分子背景は症例間で比較的均一だろう。そして、そのような希少ながんの診断法や治療法は正診率、奏効率が比較的高いと予測される[3]。肺がんのように分子背景が複雑で多種多様なサブタイプが存在するメジャーがんと対比させると、その種の希少ながんの特殊性がわかりやすい。逆に考えると、メジャーがんとは、分子背景が異なりつつ臨床像が共通しているたくさんの希少がんの集まりなのかもしれない。そのように考えると、希少がんの研究からがん研究一般につながる道筋が見えるような気がする。
特定の発がん物質が発症の原因となるがんの場合、原因となりうる物質の存在が希だと、そのがんは希少になるだろう。たとえば、悪性胸膜中皮腫はアスベストに暴露した方に多く発症するのだが、本邦では希少ながんである。「本邦では」と条件をつけたのは、悪性胸膜中皮腫の原因となる物質が多量に存在する海外の地域(カッパドキア、トルコ)では、悪性胸膜中皮腫は希少なではないからである[4]。希少である原因を考えたときに、それが希な発がん物質が原因だということがわかれば、予防法の開発ができるかもしれない。
がん細胞に対応する正常細胞の数が少なかったり、発がんに抵抗性をもっていたりする場合にも、そのがん種は希少になりうる。たとえば、眼腫瘍は希少だが、それはがんになりうる正常細胞の数が少ないからなのかもしれない。心臓のがんも希少だが、それはがんになりうる心臓の細胞(心筋細胞や血管細胞)があまり分裂せず遺伝子の変異が発生する頻度が低いため、発がんに抵抗性を示すからだろう。
原因となる遺伝子が特定できず共通の分子機構がわからなかったり、なぜ希少なのか見当がつかないという希少ながんの方が多いだろう。例えば上述の肉腫の例でいえば、融合遺伝子など特有の遺伝子異常が知られていない肉腫は多く、それらの肉腫はなぜ希少なのか、また別の理由があるのだろう。
希少がんの特殊性について述べてきたが、希少がんの研究と一般的ながん研究との間で共通する点は数多くある。予防、診断、治療というがん研究の普遍的な研究テーマは希少がんの研究においても当然ながら重要である。臨床に役立つ成果を目指すという意味では、希少がんの研究はメジャーがんの研究と本質的に同じである。使用するサンプルも腫瘍組織や血液と同じであり、実験の手法も同じものを使うことができる。ただし、症例数が少ないことによる特殊事情は存在する。たとえば、症例数が極端に少ないので検診には向かない、市場規模が小さいので製薬企業があまり関心を示さない、など。このような希少がんの特殊問題につながる方法論の開発を、個々の希少ながんごとに個別的に行うのではなく広く試みること、そのための方法論を開発したり、研究プラットフォームを構築することが、希少がん研究のユニークな点である。
具体的には、たとえば、希少がんにおいては、既存の薬剤の適応拡大はとりわけ現実的だと考えられる。希少がんでは市場規模が極端に限られており、新しい抗がん剤を一から開発することは見込まれる利潤からして難しいかもしれない。一方、浸潤や転移を来す分子機構はおそらくあらゆるがん種である程度は共通なので、すでに他のがんで承認されている抗がん剤が奏効する希少ながんは多いと考えられる。筆者の研究室では、FDA承認された抗がん剤のライブラリーを使って希少ながんの細胞株をスクリーニングしている。そして、意外にも希少ながんに奏効する抗がん剤がいくつも存在することを見いだしている(図2)。このように、既存薬の適応拡大のための実験プラットフォームを整え、臨床試験までの道のりを確立することは、希少がん研究の重要な課題である。
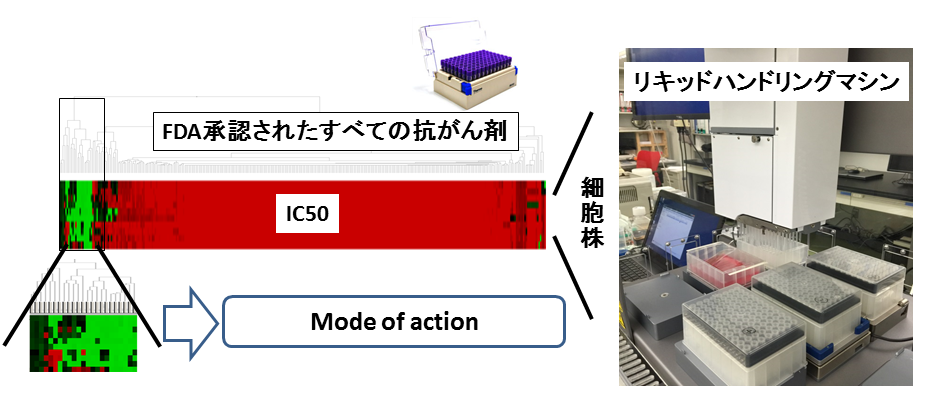
図2:適応拡大に向けたスクリーニング。既存の抗がん剤のうち希少がんへの適応拡大に利用可能なものを同定している。同定した後は薬効の分子背景を調べている。
希少がんでは臨床検体を用いた研究は一般に困難である。バイオバンクを充実させることが一つの解決法だが、仮にすばらしいバイオバンクができたところで、蓄えることができる試料は数・量ともに限られている。研究が盛んになれば供給が追い付かないので試料はすぐに枯渇してしまう。そうそう簡単に研究が完遂するとは思えず、新しい解析手法は次々と現れてくる。したがって、バイオバンクを構築するだけでは、本質的には希少ながんの臨床検体の問題の解決にならないだろう。ここで考え方を変えてみると、我々が必要なのは臨床検体そのものではなく臨床検体から得られる情報である。ゲノム、トランクスリプトーム、プロテオーム、メタボロームといったオミクス情報を臨床検体から抽出し、多くの研究者が有効に活用できる研究基盤を構築することで、「臨床検体の数が限られている」という希少がん特有の問題にアプローチすることができる。すなわち、「有限の試料(臨床検体)を無限の試料(遺伝子・タンパク質データ)に変換する」のである。その意味で、データベースの整備は希少がん研究ではとりわけ重要である。また、既存のデータの再利用も希少がんではとりわけ重要である。希少ながんとは言え、たいていの場合は誰かが実験(たとえば網羅的解析)をある程度は実施している。残念なことに個別的に少数のサンプルで実験が行われており、それぞれの研究からはちゃんとした結論が導き出せていないことも多い。しかし、幸いなことに多くの場合、網羅的解析のデータはGene Expression Omnibusなどの公的なデータベースに登録されている。このような公的データの活用が、希少がん研究ではとりわけ重要である。筆者のラボでは膨大な数の論文を精読し、実験に使える質のデータを整理して使っている。一度に数1000症例の希少ながんの発現データを使った検証実験が可能である。さらにデータベースを充実させることで、「臨床検体が得難い」という希少がん研究の問題点を、かなり解決できそうである。網羅的解析の実験プラットフォームが標準化されている場合はよいのだが、プロテオーム解析のように手法やデータフォーマットが統一されていない場合は難しい。この難しさは希少がん研究に特有のものではなく、いろいろな局面で共有される問題である。希少がんの研究としての取り組みを通じて、いつか近い将来に解決されるのではないかと期待している。
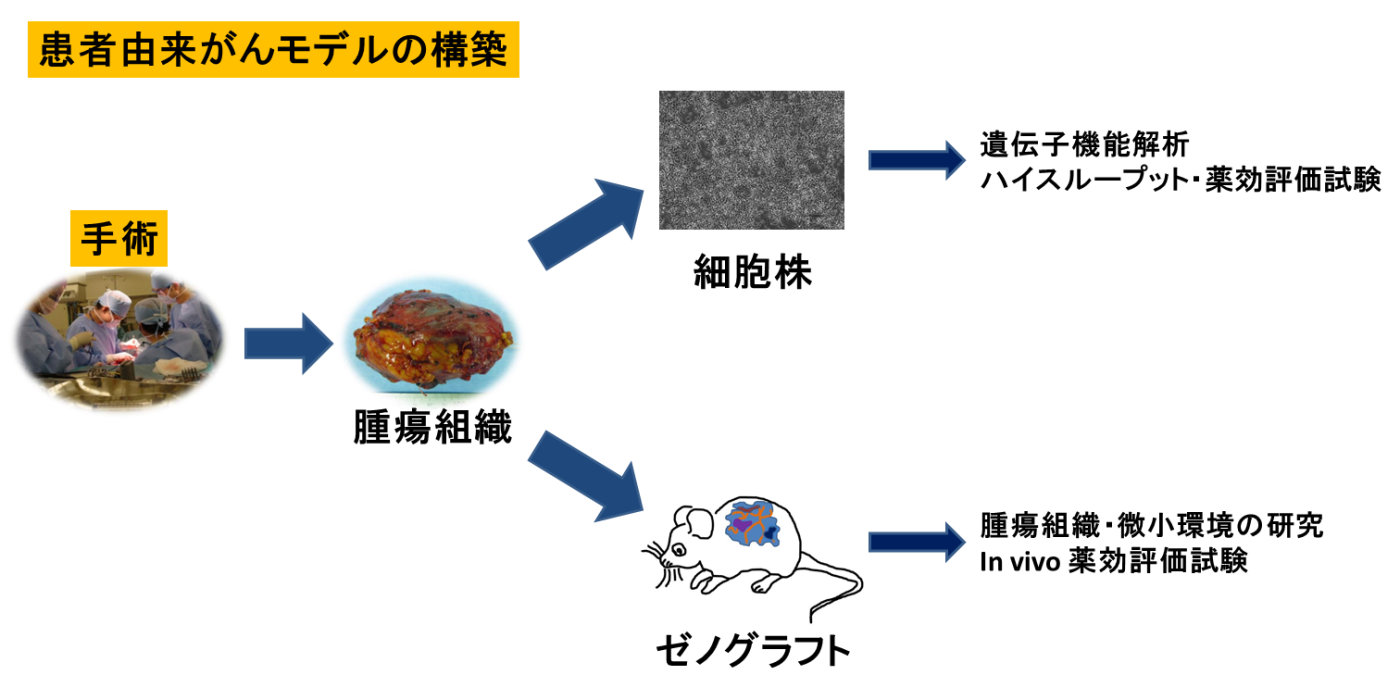
Patient-derived cancer modelすなわち臨床検体から得られるゼノグラフト、初代培養細胞、細胞株といったリソースの開発はがん研究では重要である。標準的に使われている治療法の開発も知られている分子機構の解明も、モデル系なかりせばありえなかっただろう。しかしながら、がんのモデル系は希少がんではきわめて得難い[5]。そしてそのことが研究開発が進まない一因となっている。Patient-derived cancer modelは増やして配布することも可能なので、バイオバンクの基本的な問題の解決になりうる。すなわち、「有限の試料(臨床検体)を無限の試料(patient-derived cancer model)に変換する」ことで、「臨床検体の数が限られている」という希少がん特有の問題にアプローチすることができる。筆者の研究室では、年間50-70例の肉腫症例から腫瘍組織を得てpatient-derived cancer model を樹立している。すでに40症例以上においてxenograft株や細胞株の樹立に成功しており、上述の抗がん剤ライブラリーのスクリーニングにも使用している。元の腫瘍との相違点を調べることで、実験モデルの最適な使用法をみいだせるのではないかと考えている。プロテオームを調べてみると、予想通りモデルができる過程で分子背景は変化している。何にでも使えるオールマイティーなモデル系というのは存在しなさそうである。網羅的遺伝子・タンパク質のデータを参考に、研究テーマに合ったモデルを選択したり作製したりする必要があるだろう。今までに樹立したモデル(patient-derived cancer model)は新しい治療法の開発に多くの方に使用していただきたいと考えている。今のところ作製しているのは肉腫モデルだけなのだが、筆者自身は特定のがん種に拘りはなく、希少がんにも拘らず、機会があればいろいろながんを対象に研究をしたいと考えている。新しい抗がん剤をお持ちの方でpatient-derived cancer modelを用いた実験に関心のある方は、ぜひご連絡いただきたい。
研究の手法や新しい技術は、いわゆるメジャーがんでまずしっかり開発され試されてから、やがて希少がんにも応用される、という流れが一般的である。その逆の流れ(リバース・イノベーション)が可能ではないかと私は考えている。たとえば、ここまででご紹介した研究手法は、希少ながんを対象として開発し応用しているが、発生頻度の高いメジャーながんにも十分に応用可能である。適応拡大も、データベース構築も、patient-derived cancer modelの作製も、あらゆるがんで必要とされている。また、メジャーながんとは言え、層別化を進めていくと個々のがんはやがては希少ながんになってしまうので、希少がん研究の考え方は広く一般に通じるものがある。希少がんは研究開発に適した研究対象かもしれないと考えている。なぜならば、ある種の希少がんは分子背景が均一で、少ないサンプル数からでも意味のある結果を得ることができるかもしれないからである。検体の数が少ないという問題は希少がん研究に特有ではなく、メジャーがんでも普遍的に認められる。ただ、メジャーがんの場合は「検体が得難い」というのはそれぞれの研究者の個人的な研究環境の問題であるのに対して、希少がん研究の場合は個々の研究者の問題ではなく研究分野全体の問題である。したがって、検体が少ないときの有効なアプローチの開発は、メジャーがんよりも希少がんにおいて取り組み易いのではないだろうか。例えば、「肺がん研究において検体が少ないときの解析方法の工夫」というのは研究課題としてぴんと来ないが、「希少がん研究において検体が少ないときの工夫」というのであれば、広く共有される重要な課題だ、と思われるのではないだろうか。「今の研究を希少ながん以外のがん種に応用するにはどうしたらよいか」という視点を常に持つことで、リバース・イノベーションが可能になるだろう。
4.肉腫のプロテオーム解析
筆者は、希少がん研究分野を立ち上げる前は、プロテオーム解析を用いてさまざまながんの研究を行ってきた。レーザーマイクロダイセクションのアプリケーション[6]や大型のゲルを用いた蛍光二次元電気泳動システムを構築し[7, 8]、肺がん、大腸がん、胃がん、肝がん、膵がん、胆道がん、食道がん、皮膚がん、腎がん、など一般的ながんに加え、肉腫を研究対象としてきた(図3)。
図3:臨床検体をもちいたがんプロテオーム解析。レーザーマイクロダイセクションを用いて、腫瘍組織から必要な細胞を回収する。回収した細胞からタンパク質を抽出し、プロテオーム解析で発現プロファイルを作成する。重要な臨床事象(奏効性、転移、再発など)に発現相関するタンパク質を特定し、その臨床的意義を検証する。最終的には臨床検査などの形で臨床に還元することを目指す。
なぜこんなにいろいろながん種を対象にしてきたかといえば、私が特定のがん種に拘らず何でも興味をもつタイプの研究者だということと、研究環境によるところが大きい。私は医学部を卒業した直後からタンパク質の発現解析を始め、プロテオミクスの解析技術を用いた研究を行ってきた。臨床経験がないので、特定のがん種に拘る理由がなかった。10年ほど前から国立がん研究センターで研究室を主宰してきたのだが、私の研究室で実験を行うのはほとんどが卒後10年以内の若手臨床医だった。診療を2-3年中断してトランスレーショナル・リサーチに従事したいという熱い若手臨床医を受け入れて研究を行ってきたのである。プロテオーム解析としてはすることは同じなので、研究をしたいという若手臨床医の興味にしたがってがん種を決めていた。その結果として、いろいろながんを研究対象にすることになった。どのがんもそれぞれに臨床的な課題があっておもしろく、プロテオミクスで何ができるかをがん種ごとに考え、必要に応じて技術開発もするという、なかなか楽しい日々を過ごしてきた。それぞれのがん種の特殊問題がある一方で、あらゆるがん種に共通の重要テーマもあり、俯瞰的にがん研究を眺めるという、今にして考えると、希少がん研究を始めるためのまたとない機会を得ていたのである。
整形外科・骨軟部腫瘍科からも2年ごとに新しく若手臨床医が研究に参加してきており、彼らが専門とする肉腫を対象としたプロテオーム解析を行ってきた。プロテオーム解析で肉腫研究として何ができるのかということで、先ず初めに着手したのが、組織型に関わるタンパク質の同定である。いろいろな組織型の肉腫に特徴的なタンパク質群を、プロテオーム解析で同定した(図4)[9]。顕微鏡でみて明らかに形態が異なる腫瘍であれば、組織型に対応するタンパク質は必ずみつかることを、後のプロテオーム解析で繰り返し経験することになる。しかし当時は、プロテオーム解析の期待以上のパフォーマンスにとても興奮したことを覚えている。
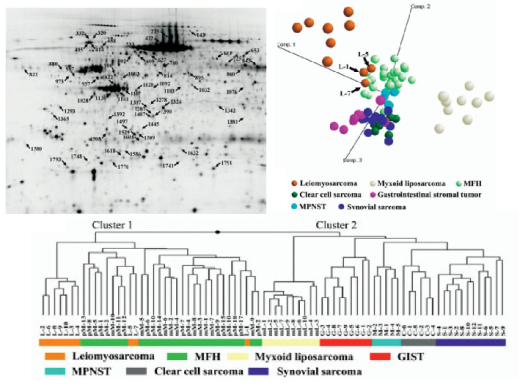
図4:蛍光二次元電気泳動法のデータの解析。蛍光二次元電気泳動法を用い(左上)、異なる組織型の肉腫に対応したタンパク質発現プロファイルを同定した(右上、下)。
今ではプロテオームのデータをバイオインフォマティクスの手法で解析するというのは、ごく一般的である。しかし2000年代初頭はほとんど行われていなかった。当時はプロテオーム解析のソフトには統計解析の機能はほとんど実装されておらず、数値データを画像解析ソフトの外に出すことすらできなかった。今となっては隔世の感がある。DNAマイクロアレイの論文を横目に、どのような手法がプロテオーム解析に使えるかを検討し、バイオマーカー解析に有用なアプローチを確立した。後に独立して研究室を主宰することになったときに、その種の研究をさらに発展させるために研究室の名称を「プロテオーム・バイオインフォマティクス・プロジェクト」とした。
ある程度は深く技術を開発したあとで、横に広く研究を拡張して行ったのが各種のがんのバイオマーカーの開発である。メジャーがん(肺がん、大腸がん、肝細胞癌、膵がん、胃がんなど)に平行して肉腫の解析も進めた。骨肉腫[10, 11]、Ewing肉腫[12, 13]、消化管間質腫瘍[14]、滑膜肉腫[15, 16]、類上皮肉腫[17(PDF:3.3MB)]、と、いろいろな肉腫のプロテオームを調べ、バイオマーカーの候補タンパク質を同定した。独立した症例での免疫染色を用いた検証実験は、プロテオーム解析以上に難しかったが、病理部の方々のご指導により、きちんとしたデータを得ることができた。消化管間質腫瘍は、予後予測バイオマーカー・フェチンを同定し、その後の多施設共同研究で予後予測能を検証することができた(図5)。バイオマーカー以外にも、骨肉腫ではスフェロイド形成に伴う化学療法抵抗性に関わるタンパク質を同定し[18]、胞巣状軟部肉腫では適応拡大につながる抗がん剤を同定することができた[19]。
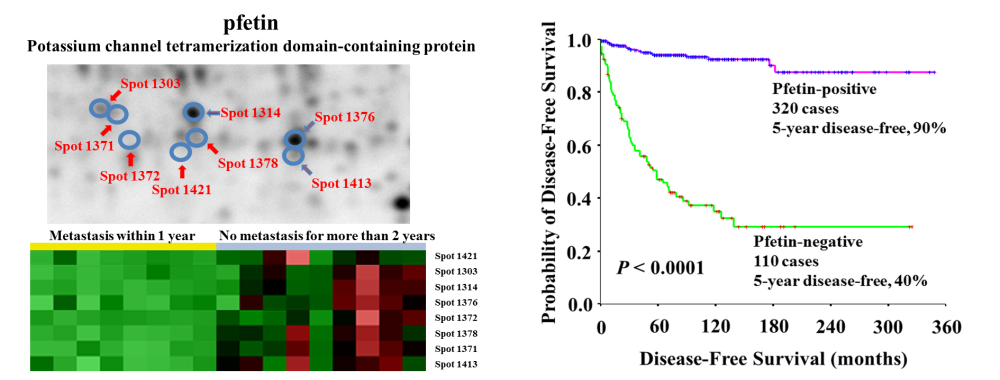
図5:消化管間質腫瘍の予後予測バイオマーカー・フェチンの例。蛍光二次元電気泳動法ではフェチンは複数のスポットで観察される。観察されるスポットのすべてが、予後不良症例で濃度が減少していた(左図)。免疫染色を用いた検証実験で、フェチンの発現が高い症例の予後は有意に良好であることを確認した(右図)。
プロテオームデータのデータベースの構築にも着手し、Genome Medicine Database of Japan Proteomics(GeMDBJ Proteomics)という、蛍光二次元電気泳動法を基盤としたオープン・データベースを構築した[20, 21]。肺がん[22]や肝細胞癌のようなメジャーながんに加え、Ewing肉腫のデータも同データベースに格納した[23]。がんのプロテオームのデータベースとしては、構築した当時としては世界最大規模だった。データベースの構築を通じて、プロテオームとトランスクリプトームの融合も意識するようになった。そして、公的データベースのmRNA発現データを用いたり[13, 24]、プロテオームのデータをトランスクリプトームのデータと統合して解析したりすることで[25]、希少がん研究の困難な点を解決できるかもしれないと考えるようになった。ここでもバイオインフォマティクスは大活躍。プロテオームの解釈はバイオインフォマティクスなしには不可能だと思う。
プロテオーム解析以外にもトランスクリプトーム解析にも着手し、骨肉腫の術前化学療法の奏功性を予測するバイオマーカーとして有望なマイクロRNAを同定した(図6)[26]。骨肉腫では術前化学療法の奏効性が予後に大きな影響を与える。化学療法に抵抗性を示す骨肉腫に特徴的なmiRNAを同定した。特定のmiRNAの発現が高い腫瘍細胞は化学療法に抵抗性を示すことを、独立した症例群やin vitro実験で証明することができた。
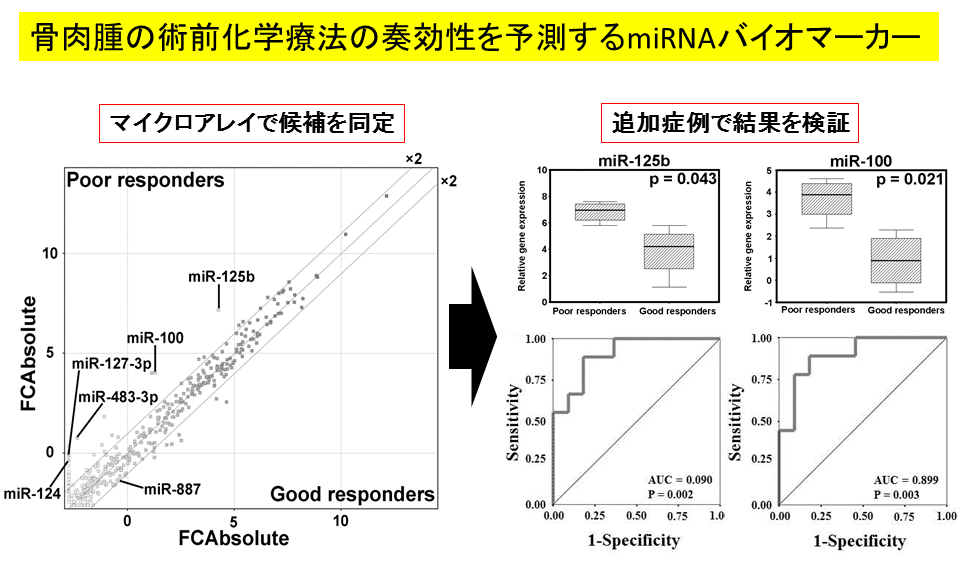
図6:骨肉腫の術前化学療法の奏効性を予測するバイオマーカー。miR-125bとmiR-100が過剰発現していると化学療法に抵抗性を示すことがわかった。
一連のプロテオーム解析の経験を通じて痛感したのが、肉腫の培養細胞が手に入らなくて研究が進まないこと、バイオバンクがきちんと整備されておらず実験が最初から難航すること、メタ解析のための大規模な公的なデータベースが必要であること、などである。「希少がん研究分野」を主宰することになったときに、このような経験を活かして希少がん研究の方向性を決めることになった。肉腫のプロテオーム解析を行っているときには、自分が希少がんを標榜する研究室を立ち上げることになるとは思ってもみなかった。人生おもしろいものである。
5.最後に
希少がんを研究するとは、特定の種類の希少がんを研究するということだけでなく、希少がん全般にわたる普遍的な課題を根本的に解決すること、そして希少がんの問題の解決のためのプラットフォームを開発することだと考えている。そのような考えのもとに希少がん研究の基盤構築に取り組んでいる。同時に、個々の希少がんを研究し、臨床に還元できる成果を短期間で得ることも重要である。両者はどちらか一方だけでよいというものではなく、お互いにフィードバックをかけて並行して進めなくてはいけない。希少がん研究分野の設立より前に開発したバイオマーカーの実用化を進めており、そこで培ったノウハウをシステム化したいと考えている。さらに、希少がん研究で構築したシステムを、やがてはがん研究一般に応用できるようにすること(「リバース・イノベーション」)も重要である。本稿では筆者の研究の背景と現在の取り組みをご紹介した。
研究の道に入ったばかりのころは、研究とは自分が楽しければそれでいいのだと考えていた。今となっては、かつてそのような狭い料簡で職業を選択したことが信じがたい気がする。今の職場で働くようになり、国内外のいろいろな分野の方々と出会い、我々の研究があってよかったと、患者さん、医療従事者、そして世界中の方々に思っていただける研究成果を目指すようになった。これまでの自分の変遷を考えると今の研究テーマや考え方が一生続くのかどうか正直わからないのだが、研究はおもしろい仕事だという思いは変わることない。研究職というものが存在しない時代に生まれていたら何をしていただろうか。この時代に生まれ、この職業に出会うことができた僥倖を味わいながら、悔いのないように精一杯がんばりたいものだと思う今日この頃である。
6.文献
- Gatta G, van der Zwan JM, Casali PG, Siesling S, Dei Tos AP, Kunkler I, Otter R, Licitra L, Mallone S, Tavilla A et al: Rare cancers are not so rare: the rare cancer burden in Europe. Eur J Cancer 2011, 47(17):2493-2511.
- Tamaki T, Dong Y, Ohno Y, Sobue T, Nishimoto H, Shibata A: The burden of rare cancer in Japan: application of the RARECARE definition. Cancer Epidemiol 2014, 38(5):490-495.
- Braiteh F, Kurzrock R: Uncommon tumors and exceptional therapies: paradox or paradigm? Mol Cancer Ther 2007, 6(4):1175-1179.
- Carbone M, Emri S, Dogan AU, Steele I, Tuncer M, Pass HI, Baris YI: A mesothelioma epidemic in Cappadocia: scientific developments and unexpected social outcomes. Nat Rev Cancer 2007, 7(2):147-154.
- Pan X, Yoshida A, Kawai A, Kondo T: Current status of publicly available sarcoma cell lines for use in proteomic studies. Expert Rev Proteomics 2016, 13(2):227–240.
- Kondo T, Seike M, Mori Y, Fujii K, Yamada T, Hirohashi S: Application of sensitive fluorescent dyes in linkage of laser microdissection and two-dimensional gel electrophoresis as a cancer proteomic study tool. Proteomics 2003, 3(9):1758–1766.
- Kondo T, Hirohashi S: Application of highly sensitive fluorescent dyes (CyDye DIGE Fluor saturation dyes) to laser microdissection and two-dimensional difference gel electrophoresis (2D-DIGE) for cancer proteomics. Nature Protocols 2007, 1(6):2940-2956.
- 近藤格: DIGE道場. 東京: ライフアカデミーサイエンス; 2013.
- Suehara Y, Kondo T, Fujii K, Hasegawa T, Kawai A, Seki K, Beppu Y, Nishimura T, Kurosawa H, Hirohashi S: Proteomic signatures corresponding to histological classification and grading of soft-tissue sarcomas. Proteomics 2006, 6(15):4402-4409.
- Kikuta K, Tochigi N, Saito S, Shimoda T, Morioka H, Toyama Y, Hosono A, Suehara Y, Beppu Y, Kawai A et al: Peroxiredoxin 2 as a chemotherapy responsiveness biomarker candidate in osteosarcoma revealed by proteomics. Proteomics Clin Appl 2010, 4(5):560-567.
- Kubota D, Mukaihara K, Yoshida A, Tsuda H, Kawai A, Kondo T: Proteomics study of open biopsy samples identifies peroxiredoxin 2 as a predictive biomarker of response to induction chemotherapy in osteosarcoma. J Proteomics 2013, 91:393-404.
- Kikuta K, Tochigi N, Shimoda T, Yabe H, Morioka H, Toyama Y, Hosono A, Beppu Y, Kawai A, Hirohashi S et al: Nucleophosmin as a candidate prognostic biomarker of Ewing's sarcoma revealed by proteomics. Clin Cancer Res 2009, 15(8):2885-2894.
- Haga A, Ogawara Y, Kubota D, Kitabayashi I, Murakami Y, Kondo T: Interactomic approach for evaluating nucleophosmin-binding proteins as biomarkers for Ewing's sarcoma. Electrophoresis 2013, 34(11):1670-1678.
- Suehara Y, Kondo T, Seki K, Shibata T, Fujii K, Gotoh M, Hasegawa T, Shimada Y, Sasako M, Shimoda T et al: Pfetin as a prognostic biomarker of gastrointestinal stromal tumors revealed by proteomics. Clin Cancer Res 2008, 14(6):1707-1717.
- Suehara Y, Tochigi N, Kubota D, Kikuta K, Nakayama R, Seki K, Yoshida A, Ichikawa H, Hasegawa T, Kaneko K et al: Secernin-1 as a novel prognostic biomarker candidate of synovial sarcoma revealed by proteomics. J Proteomics 2011, 74(6):829-842.
- Suehara Y, Kikuta K, Nakayama R, Tochigi N, Seki K, Ichikawa H, Fujii K, Hasegawa T, Shimoda T, Kurosawa H et al: GST-P1 as a histological biomarker of synovial sarcoma revealed by proteomics. Proteomics - Clinical Applications 2009, 3(5):623-634.
- Mukaihara K, Kubota D, YOshida A, Asano N, Suehara Y, Kaneko K, Kawai A, Kondo T: Proteomic profile of epithelioid sarcoma. J Proteome Biioinform 2014, 7(7):158-165.
- Arai K, Sakamoto R, Kubota D, Kondo T: Proteomic approach toward molecular backgrounds of drug resistance of osteosarcoma cells in spheroid culture system. Proteomics 2013, 13(15):2351-2360.
- Kubota D, Yoshida A, Kawai A, Kondo T: Proteomics identified overexpression of SET oncogene product and possible therapeutic utility of protein phosphatase 2A in alveolar soft part sarcoma. J Proteome Res 2014, 13(5):2250-2261.
- Genome Medicine Database of Japan Proteomics(GeMDBJ Proteomics) [https://gemdbj.nibio.go.jp/dgdb/DigeTop.do]
- Kondo T: Cancer proteome-expression database: Genome Medicine Database of Japan Proteomics. Expert Rev Proteomics 2010, 7(1):21-27.
- Kosaihira S, Tsunehiro Y, Tsuta K, Tochigi N, Gemma A, Hirohashi S, Kondo T: Proteome expression database of lund adenocarcinoma: a segment of the Genome Medicine Database of Japan Proteomics. J Proteome Biioinform 2009, 2(11):463-465.
- Kikuta K, Tsunehiro Y, Yoshida A, Tochigi N, Hirohahsi S, Kawai A, Kondo T: Proteome Expression Database of Ewing Sarcoma: a Segment of the Genome Medicine Database of Japan Proteomics. Journal of Proteomics & Bioinformatics 2009, 02(12):500-504.
- Kubota D, Yoshida A, Tsuda H, Suehara Y, Okubo T, Saito T, Orita H, Sato K, Taguchi T, Yao T et al: Gene expression network analysis of ETV1 reveals KCTD10 as a novel prognostic biomarker in gastrointestinal stromal tumor. PLoS One 2013, 8(8):e73896.
- Ichikawa H, Yoshida A, Kanda K, Kosugi S, Ichikawa T, Hanyu T, Taguchi T, Sakumoto M, Katai H, Kawai A et al: Prognostic significance of PML expression in gastrointestinal stromal tumor; integrated proteomic and transcriptomic analysis. Cancer Sci 2014, 106(1):115-124.
- Kubota D KN, Fujiwara T, Yoshida A, Arai Y, Qiao ZZ, Takeshita F, Ochiya T, Kawai A, Kondo T: miR-125b and miR-100 Are Predictive Biomarkers of Response to Induction Chemotherapy in Osteosarcoma. Sarcoma 2016, 2016(2016):Article ID 1390571.

