トップページ > 研究組織一覧 > 分野・独立ユニットグループ > 医療AI研究開発分野 > 研究プロジェクト > データサイエンス研究コア
データサイエンス研究コア
解析技術の進歩に伴い、一度に大量のオミクスデータを取得することが可能となった。臨床検体から得られるマルチオミクスデータは多くの情報を含んでおり、診療情報と組み合わせた統合解析を行うことで、様々なクリニカルクエスチョンや病態解明へとつながる潜在特徴量を抽出することが可能である。しかし、臨床検体から得られるオミクスデータは個人差やサンプル間のデータ分布のばらつきが大きく、解析結果の堅牢性や意味解釈性を担保することが困難である。そこで我々は、堅牢性と解釈性の高いマルチオミクスデータ解析手法を開発することで、病態解明や臨床応用可能な予後予測モデルの開発研究へと繋げることを目指している。
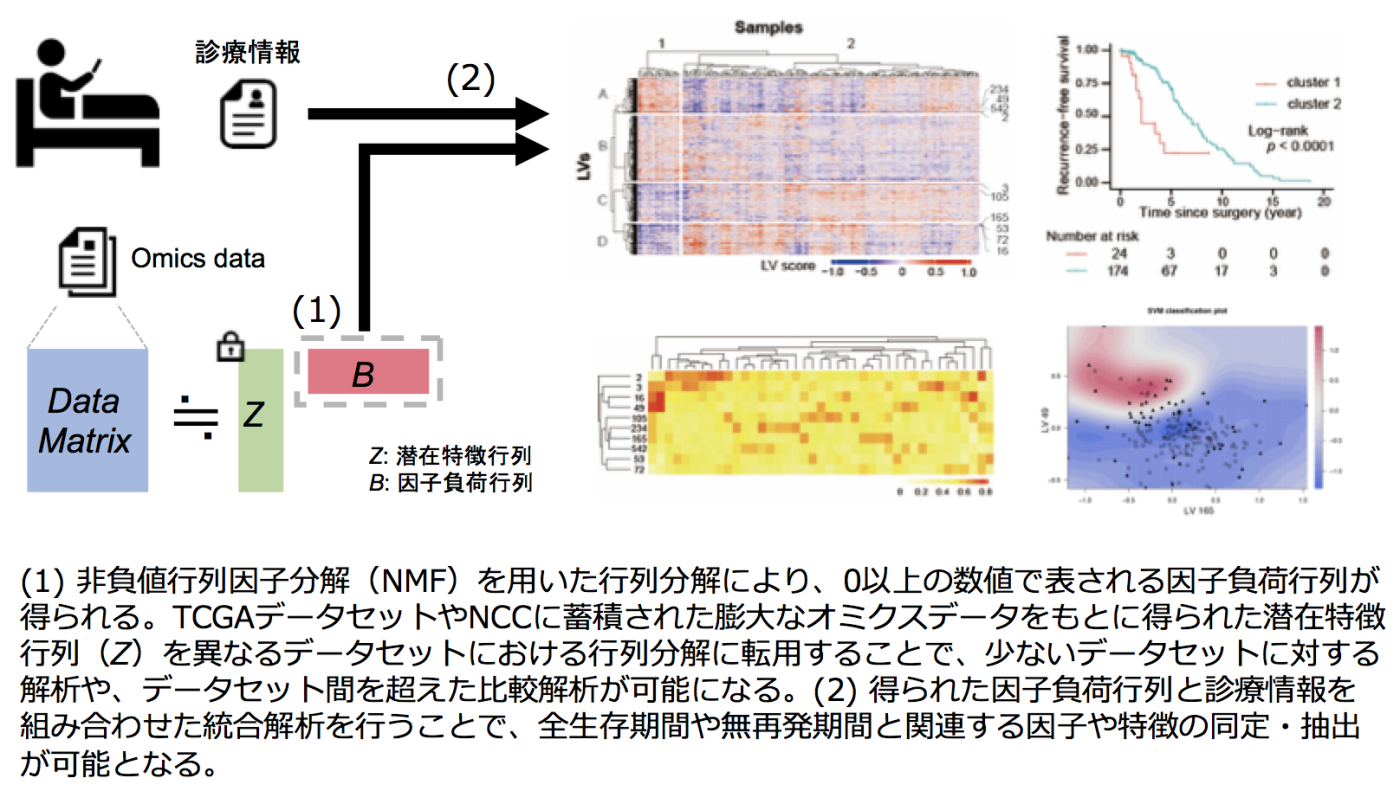
臨床検体から得られるマルチオミクスデータの多くは、検体×解析項目からなるデータ行列で表され、これらデータ行列を行列分解することで、データ中に含まれる様々な潜在特徴変数を抽出することが可能である。非負値行列因子分解(Non negative matrix factorization: NMF)では、行列分解後の潜在特徴行列と因子負荷行列がいずれも正の値を取るため、解析結果の解釈が容易であり、リアルワールドデータ解析に有用である。また、解析対象となる母集団が含まれる大規模なデータセットに対して、NMFを用いて得られた潜在特徴行列は、同一母集団から発生したと考えられる異なるデータセットに対しても有用であるため、転移学習が可能である。現在我々は、The Cancer Genome Atlas(TCGA)から得られる約1万件のDNAメチロームデータセットに対し、知識行列を用いたスパース正則化付きNMFを行う転移学習手法の開発を進めている。本解析手法によって得られた因子負荷行列を、臨床情報と組み合わせて解析することで、全生存期間や無再発期間の推定や、薬剤反応性に寄与する因子など、様々なクリニカルクエスチョンに対する洞察を得ることが可能となると考えている。
また中央病院の診療科と共同し、これらの行列分解やベイズ統計学的手法を用いた関係データ解析を、日常診療で蓄積される膨大な臨床情報に対して行うことで、リンパ節転移の予測モデルの構築や無再発生存期間予測モデル構築など、臨床的課題解決に向けたデータ解析も進めている。