トップページ > 研究組織一覧 > 基盤的臨床開発研究コアセンター > 臨床ゲノム解析部門 > 技術支援内容
技術支援内容
ゲノム解析を実施する前に必要な準備
目次
- RNA サンプルの品質検定(Sample QC)
解析に必要な RNA サンプル量
全トランスクリプトーム解析プラットフォームの紹介 - シークエンスライブラリーの品質検定 ( Library QC )
- DNAサンプルの品質検定(Sample QC)
全ゲノム・エクソームシークエンス解析プラットフォームの選定
(1) RNA サンプルの品質検定(Sample QC)
研究所臨床ゲノム解析部門では、全トランスクリプトーム解析、WTS (RNA-seq) を実施しています。RNA の質や量によって、プラットフォームを選択しているため、RNA サンプルの品質検定の評価は重要です。
手順は下記の通りに進めています。
依頼された連結組織標本より抽出した RNA の濃度と品質の確認
RIN値 と DV200 を Agilent TapeStation または Bioanalyzer を用いて確認しています。詳しくは下記のサイトをご覧ください。
https://www.chem-agilent.com/contents.php?id=1003805
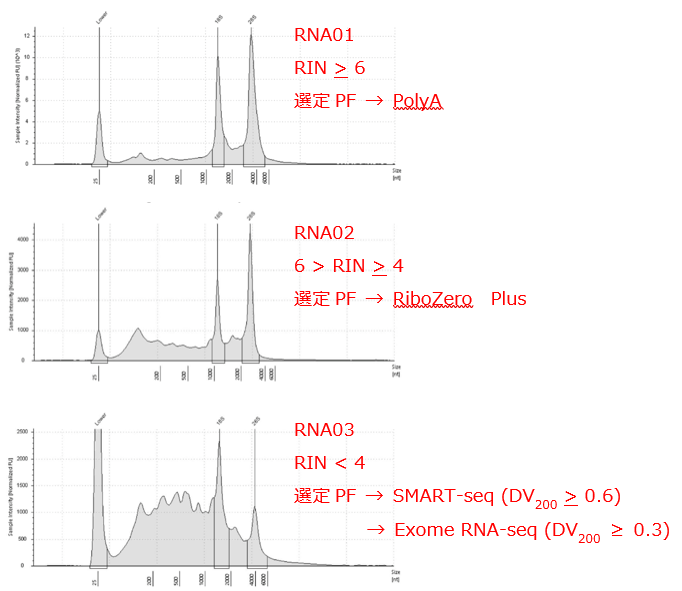
解析に必要なRNA量や品質で解析プラットホームを選定
ただし、症例間で比較する場合、統一したプラットホームを用いた解析が必要となりますのでご注意ください。
FFPE (ホルマリン固定)標本を用いた WTS (SMART-seq) 解析は可能
FFPE 由来 RNA でも、DV200 が 0.6 以上であれば SMART-seq による解析が可能です。また、0.3 以上であれば Exome RNA-seq による解析が可能です。
解析結果の返却
依頼された検体の受領から結果の返却まで 4~6 週間かかります。
◎DV200 とは、200ヌクレオチド以上のRNA断片の割合のことをいいます。詳しくは下記のサイトをご覧ください。
https://www.agilent.com/en/promotions/dv200-determination 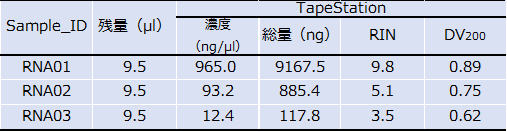
解析に必要な RNA サンプル量
解析委託時に必要となる RNA 量と品質
・PolyA RNA-seq: 4 ug以上( 200 ng/uL以上、20 uL以上、RIN値 6 以上)
→Input 量は 100 ng/uL、11 uL (1.1 ug)となります。
・RiboZero Plus RNA-seq: 2 ug以上( 80ng/uL以上、25 uL以上、RIN値 4 以上)
→Input 量は 50 ng/uL、 10 uL ( 500 ng )となります。
◎ただし、100 ngまで全量をinput量とすることで、解析を実施可能です。しかしながら、ライブラリQCで基準を満たさない場合は、シークエンスのランは行いません。
・SMART-seq: 50 ng以上( 10 ng/uL 以上、10 uL以上、DV200 0.6 以上 )
→Input 量は凍結由来RNA が 1.1 ng/uL、10 uL ( 10 ng )、
FFPE由来RNA (DV200 < 0.6 の場合)が 5 ng/uL、10 uL ( 50ng ) となります。
◎RIN値が 4 以下の場合は、SMART-seq を推奨します。
・Exome-RNA-seq
DV200 が0.6以下であっても、Exome RNA-seqによる解析が可能な場合があります。個別にご相談ください。
全トランスクリプトーム解析プラットフォームの紹介
研究所臨床ゲノム解析部門では、4つの全トランスクリプトーム解析を実施しています。RNAの質や量によって、プラットフォームを選択しています。
(注意事項)解析対象集団内のプラットフォームは統一する必要があります。
★PolyA RNA-seq(高品質RNA)
Total RNAから3’末端に付加するpolyA RNAを回収し、逆転写反応で合成したcDNAをシークエンスライブラリとして、シークエンスを実施します。
〇使用する試薬並びに機器(受託企業)
・TruSeq Stranded mRNA Library Prep (Illumina)
・IDT for Illumina - TruSeq RNA UD Indexes (Illumina)
・Agilent XT-Auto System (Agilent technologies)
〇使用する試薬並びに機器(臨床ゲノム解析部門)
・TruSeq Stranded mRNA Library Prep (Illumina)
・TruSeq RNA Single Indexes Set A/B (Illumina)
下記の手順に従って、ライブラリー調製を実施しています。
Sample QC にて RNA の質が担保されていることを確認した後、PolyA + RNA を断片化し、この RNA 断片を鋳型として逆転写反応を行い、一本鎖 cDNA を合成します。この一本鎖 cDNA を鋳型として、dUTP を取り込ませた二本鎖 cDNA を合成します。得られた二本鎖 cDNA の両末端を平滑化・リン酸化処理した後、3 ′ -dA 突出処理を行い、Index 付きアダプターを連結します。アダプターを連結した二本鎖 cDNA を鋳型とし、dUTP を持つ鎖を選択的に増幅しないポリメラーゼにより PCR 増幅を行い、シーケンスライブラリーとします。 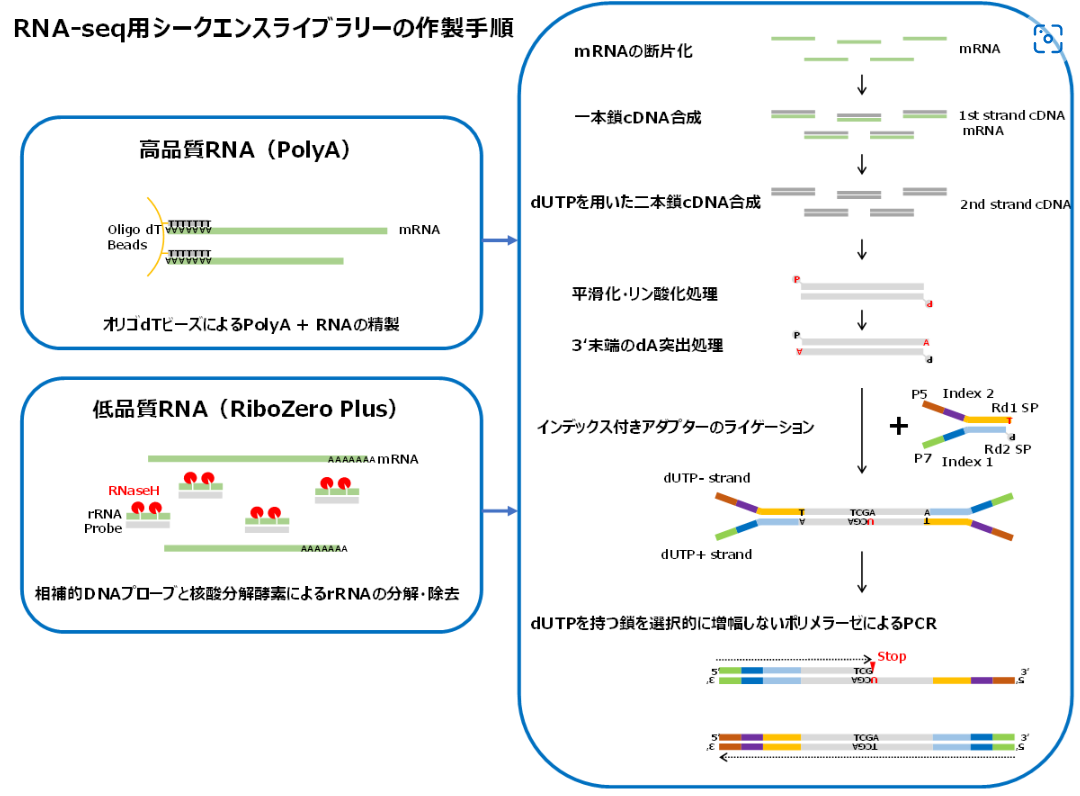
★RiboZero Plus RNA-seq(低品質RNA)
Total RNAからリボソーマルRNAを除去し、逆転写反応で合成したcDNAをシークエンスライブラリとして、シークエンスを実施します。
使用する試薬並びに機器
・TruSeq Stranded Total RNA Library Prep Gold (Illumina)
・RNAClean XP Beads (beckmancoulter)
・IDT for Illumina - TruSeq RNA UD Indexes (Illumina)
・Agilent XT-Auto System (Agilent technologies)
下記の手順に従って、ライブラリー調製を実施しています。
解析検体より rRNA を除去した後、断片化します。この RNA 断片を鋳型として逆転写反応により一本鎖 cDNA を合成します。この一本鎖 cDNA を鋳型として、dUTP を取り込ませた二本鎖 cDNA を合成します。得られた二本鎖 cDNA の両末端を平滑化・リン酸化処理した後、3 ′ -dA 突出処理を行い、Index 付きアダプターを連結します。アダプターを連結した二本鎖 cDNA を鋳型とし、dUTP を持つ鎖を選択的に増幅しないポリメラー ゼにより PCR 増幅を行い、シーケンスライブラリーとします。
★SMART-seq Stranded
Total RNAからランダムプライマーを用いてcDNAを合成した後、リボソームcDNAの切断し、シークエンスライブラリーとして、シークエンスを実施します。
使用する試薬
・ SMART-Seq Stranded Kit
・ SMARTer RNA Unique Dual Index Kit
下記の手順に従って、ライブラリー調製を実施しています。
Total RNA に対して、ランダムプライマーを用いて cDNA を合成し、SMART(Switching Mechanism At 5’ End of RNA Template)法により 1st strand cDNA の末端に特定の配列を付与します。引き続き、特定配列 を利用し、検体ごとに異なるタグ配列を有する Index 付きのプライマーを用いて PCR により増幅します(PCR:18-19サイクル)。得られた PCR 産物を、AMPure XP(ベックマン・コールター社)を用いた磁気ビーズ法にて精製し、scZapR と scR-Probe により、リボソーム cDNA の切断を行います。最後に、アダプターを認識するプライマーを使用して未切断 cDNA を PCR 増幅し、シーケンスライブラリーとします。
★Exome RNA-seq(低品質RNA)
Total RNAを逆転写して合成したcDNAから、全エクソンシークエンス用のベイトを用いて遺伝子配列をキャプチャーしてシークエンスライブラリーを作製し、シークエンスを実施します。
使用する試薬並びに機器
・SureSelect XT HS2 RNA 試薬キット (index primer pairs 1-96) (Agilent technologies)
・SureSelect XT Human All Exon V7 キャプチャライブラリ (Agilent technologies)
(2) シークエンスライブラリーの品質検定 ( Library QC )
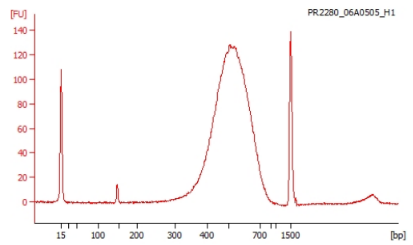
★作製したライブラリーのインサートサイズをAgilent TapeStationまたはBioanalyzerで確認します。
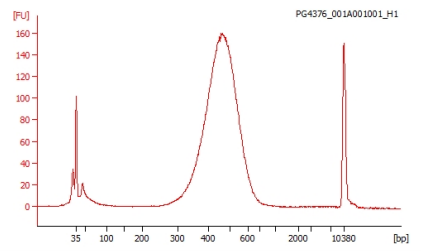
サンプルDNAの品質検定の例
作製されたシーケンスライブラリーの品質を Agilent 2100 Bioanalyzer を用いて測定します。なおライブラリーはアダプターを付加しているため、ピークサイズからアダプターサイズ(約 100base)を除いたサイズがクローニングサイズになります。
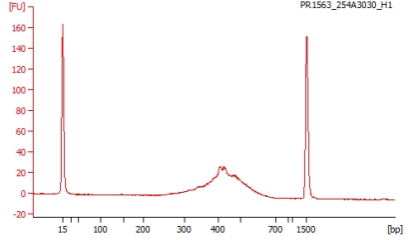

NovaSeq 6000によるシークエンス解析
物理的に数百baseに断片化したDNAからライブラリーを作製してフローセルに結合させ、Sequencing by Synthesis法による2色蛍光検出により塩基配列を一挙に解析します。NovaSeq 6000 S4試薬の場合、1フローセルあたり約2,400 Gbase、および約160億リードの塩基配列データの取得が可能です。150bp paired end sequenceを行います。
(3) DNAサンプルの品質検定(Sample QC)
研究所臨床ゲノム解析部門では、全ゲノム・エクソームシークエンス解析を実施しています。DNAの質や量によって、プラットフォームを選択しています。RNA-seqとは異なり、一般的に、異なるプラットフォームで得られたデータであっても比較解析を行うことができます。
アガロースゲル電気泳動結果の例
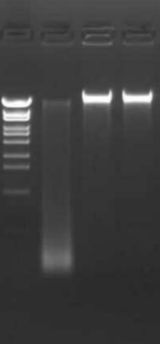
★送付された検体の二重鎖DNA濃度をThermo Fisher Qubitなどで確認します。また状況によっては、アガロースゲル電気泳動で高分子DNAを確認します。
★提供いただくDNA量は、 以下の通りです。
WGS(PCR Free): 4 ug (80 ng/uL, 50 uL以上)、2ug (80 ng/uL, 25 uL以上)
WGS(Tagmentation): 1 ug (50 ng/uL, 20 uL以上)
WES:1 ug (20 -80 ng/uL, 50uL以上)
サンプルDNAの品質検定の例
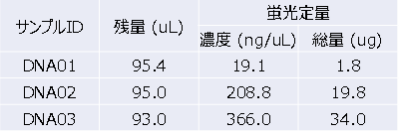
アガロースゲル電気泳動結果の例
(レーンの左から λ-EcoT14 I digest, DNA01 → 品質不良, DNA02, DNA03)
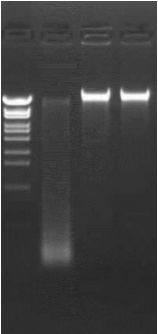
★ FFPE(Formalin-Fixed Paraffin-Embedded)由来RNAでも、WESが実施可能です。ただしDNA断片化など、ある一定のDNAサンプルの品質が担保されているかを確認する必要があります。別途ご相談をお願いします。
全ゲノム・エクソームシークエンス解析プラットフォームの選定
〇WGS( PCR Free ライブラリーの作製 )
超音波処理による物理的な断片化を行い、さらに末端平滑化後、アダプターを付けてシークエンスを行います。
・Input量:2.2 ug (40 ng /uL 55 uL) 550bp insert size
・Input量:1.1 ug (20 ng/uL 55 uL)350bp insert size
使用する試薬並びに機器
・ TruSeq DNA PCR-Free Library Prep Kit (Illumina)
・ IDT for Illumina - TruSeq DNA UD Indexes (Illumina)
・ Agilent XT-Auto System (Agilent technologies)
下記の手順に従って、ライブラリー調製を実施しています。
Sample QCをパスした検体を、アコースティックソルビライザー Covaris (コバリス社) を用いて物理的に数百 bp に断片化します。断片化した DNA の両末端 を平滑化・リン酸化処理した後、Agencourt AMPure XP(ベックマン・コ―ルター社) による DNA のサイズ選別を行います。サイズ選別された DNA に対して 3′-dA 突出末端処理を行い、index 付きアダプターを連結することで、シーケンスライブラリーとします。
★WGS(Tagmentationライブラリーの作製 )
Transposaseを用いたタグメンテーションにより断片化並びにアダプター付加を行い、シークエンスを行います。
・Input量:0.3 ug (20 ng/uL 15uL)350bp insert size
使用する試薬並びに機器
・ Illumina DNA PCR-Free Prep Kit, Tagmentation (Illumina)
・ IDT for Illumina – DNA/RNA UD Indexes (Illumina)
・ Agilent XT-Auto System (Agilent technologies)
下記の手順に従って、ライブラリー調製を実施しています。
Sample QCをパスした検体に対して、Tagmentation付加ビーズを用いて断片化した後にIndexを付加し、磁気ビーズによりサイズ選別したものをシークエンスライブラリーとします。
★WES( ライブラリーの作製 )
超音波処理による物理的な断片化を行い、さらに末端平滑化後、アダプターを付けてシークエンスを行います。
・Input量:500 ng (25 ng/uL 20 uL)
使用する試薬並びに機器
・SureSelect XT Low Input Reagents (Agilent technologies)
・SureSelect XT Low Input Dual Index P5 Indexed Adaptors1-96 (Agilent technologies) Agilent XT-Auto System (Agilent technologies)
・SureSelectXT Human All Exon Kit V7 (Agilent technologies) (約48 Mb)
・Twist Comprehensive Exome Panel (約37 Mb)
下記の手順に従って、ライブラリー調製を実施しています。
Sample QCをパスした検体をアコースティックソルビライザー Covaris (コバリス社) を用いて物理的に数百 bp に断片化します。断片化した DNA の両末端を平滑化・リン酸化処理した後、3′-dA 突出末端処理を行います。末端修飾した DNA に P5 アダプターを連結した後、Index 付きプライマーを使用して PCR による増幅を行い、DNA ライブラリーとします。
シークエンスライブラリーの品質検定(Library QC)
★作製したライブラリーのインサートサイズをAgilent TapeStationまたはBioanalyzerで確認します。
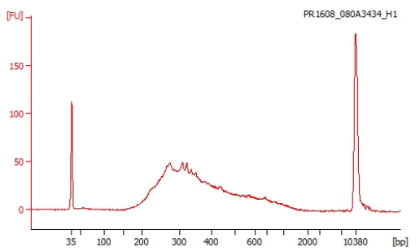
サンプルDNAの品質検定の例
作製されたシーケンスライブラリーの品質を Agilent 2100 Bioanalyzer を用いて測定します。なおライブラリーはアダプターを付加しているため、ピークサイズからアダプターサイズ(約 100base)を除いたサイズがクローニングサイズになります。
全ゲノムシークエンス用ライブラリー調製
(1)WGS(PCR Free ライブラリー)
レポート返却フォーマットの例
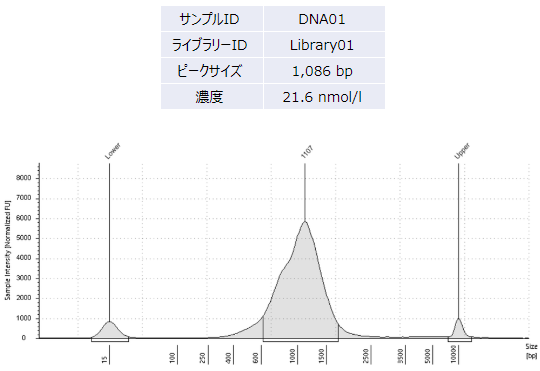
(2)WGS(Tagmentation ライブラリー)
レポート返却フォーマットの例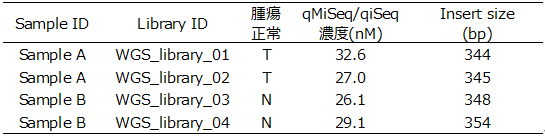
(3)WES(SureSelectXT Human All Exon Kit V7 ライブラリー)
レポート返却フォーマットの例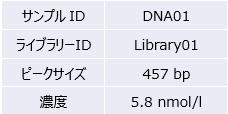
NovaSeq 6000によるシークエンス解析
物理的に数百baseに断片化したDNAからライブラリーを作製してフローセルに結合させ、Sequencing by Synthesis法による2色蛍光検出により塩基配列を一挙に解析します。NovaSeq 6000 S4試薬の場合、1フローセルあたり約2,400 Gbase、および約160億リードの塩基配列データの取得が可能です。150 bp paired end sequenceを行います。

